注記
最後の部分に移動して完全なサンプルコードをダウンロードしてください。または、Binder経由でブラウザでこの例を実行してください。
核膜での蛍光強度測定#
この例では、細胞画像の時間シーケンス(それぞれ2つのチャネルと2つの空間次元を持つ)で、核膜に局在する蛍光強度を測定するための、バイオイメージデータ解析における確立されたワークフローを再現します。この時間シーケンスは、細胞質領域から核膜へのタンパク質の再局在の過程を示しています。この生物学的応用は、Andrea Boniとその共同研究者によって最初に発表され[1]、Kota Miuraの教科書[2]やその他の研究([3], [4])でも使用されました。言い換えれば、このワークフローをImageJマクロからscikit-imageを使用したPythonに移植します。
import matplotlib.pyplot as plt
import numpy as np
import plotly.io
import plotly.express as px
from scipy import ndimage as ndi
from skimage import filters, measure, morphology, segmentation
from skimage.data import protein_transport
まず、ワークフローを構築するために、単一の細胞/核から始めます。
image_sequence = protein_transport()
print(f'shape: {image_sequence.shape}')
shape: (15, 2, 180, 183)
データセットは、15フレーム(時間点)と2チャネルを持つ2D画像スタックです。
vmin, vmax = 0, image_sequence.max()
fig = px.imshow(
image_sequence,
facet_col=1,
animation_frame=0,
zmin=vmin,
zmax=vmax,
binary_string=True,
labels={'animation_frame': 'time point', 'facet_col': 'channel'},
)
plotly.io.show(fig)
まず、最初の画像の最初のチャネルを考慮しましょう(下の図のステップa))。
image_t_0_channel_0 = image_sequence[0, 0, :, :]
核の縁のセグメンテーション#
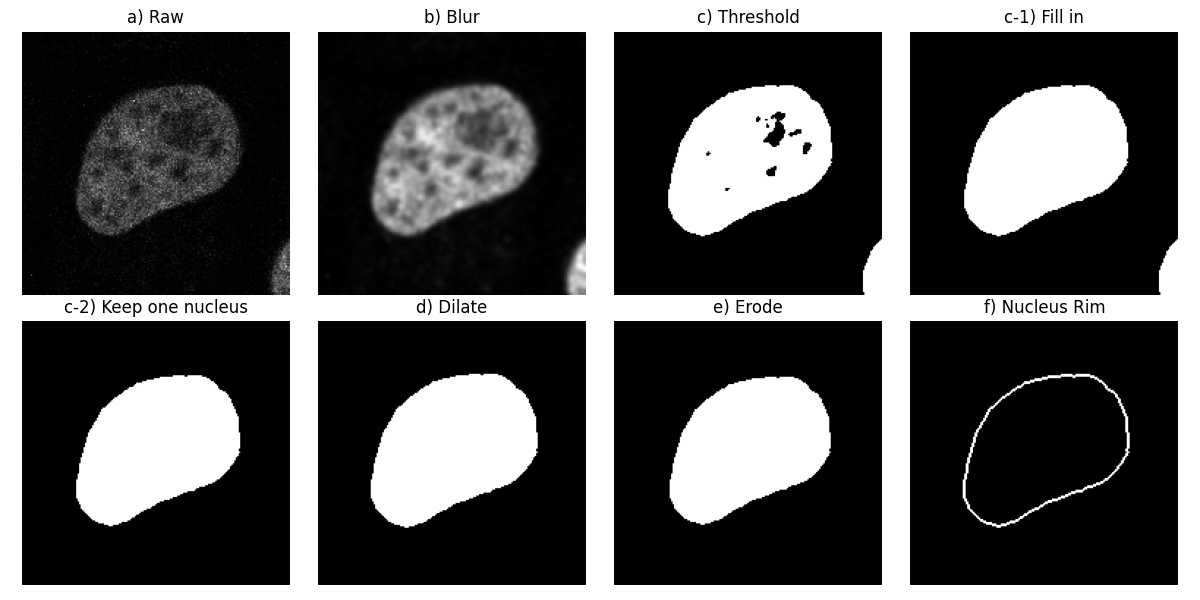
この画像を平滑化するために、ガウスローパスフィルターを適用しましょう(ステップb))。次に、大津の方法を使用して、バックグラウンドとフォアグラウンドの間の閾値を見つけて核をセグメンテーションします。これにより、バイナリ画像を取得します(ステップc))。次に、オブジェクト内の穴を埋めます(ステップc-1))。
smooth = filters.gaussian(image_t_0_channel_0, sigma=1.5)
thresh_value = filters.threshold_otsu(smooth)
thresh = smooth > thresh_value
fill = ndi.binary_fill_holes(thresh)
元のワークフローに従って、画像の境界に触れるオブジェクトを削除しましょう(ステップc-2))。ここでは、別の核の一部が右下の隅に触れていることがわかります。
dtype('bool')
このバイナリ画像のモルフォロジー膨張(ステップd))とそのモルフォロジー侵食(ステップe))の両方を計算します。
最後に、核の縁を取得するために、膨張した画像から侵食した画像を減算します(ステップf))。これは、dilateにあり、erodeにはないピクセルを選択することと同じです。
mask = np.logical_and(dilate, ~erode)
これらの処理ステップをサブプロットのシーケンスで視覚化しましょう。
fig, ax = plt.subplots(2, 4, figsize=(12, 6), sharey=True)
ax[0, 0].imshow(image_t_0_channel_0, cmap=plt.cm.gray)
ax[0, 0].set_title('a) Raw')
ax[0, 1].imshow(smooth, cmap=plt.cm.gray)
ax[0, 1].set_title('b) Blur')
ax[0, 2].imshow(thresh, cmap=plt.cm.gray)
ax[0, 2].set_title('c) Threshold')
ax[0, 3].imshow(fill, cmap=plt.cm.gray)
ax[0, 3].set_title('c-1) Fill in')
ax[1, 0].imshow(clear, cmap=plt.cm.gray)
ax[1, 0].set_title('c-2) Keep one nucleus')
ax[1, 1].imshow(dilate, cmap=plt.cm.gray)
ax[1, 1].set_title('d) Dilate')
ax[1, 2].imshow(erode, cmap=plt.cm.gray)
ax[1, 2].set_title('e) Erode')
ax[1, 3].imshow(mask, cmap=plt.cm.gray)
ax[1, 3].set_title('f) Nucleus Rim')
for a in ax.ravel():
a.set_axis_off()
fig.tight_layout()
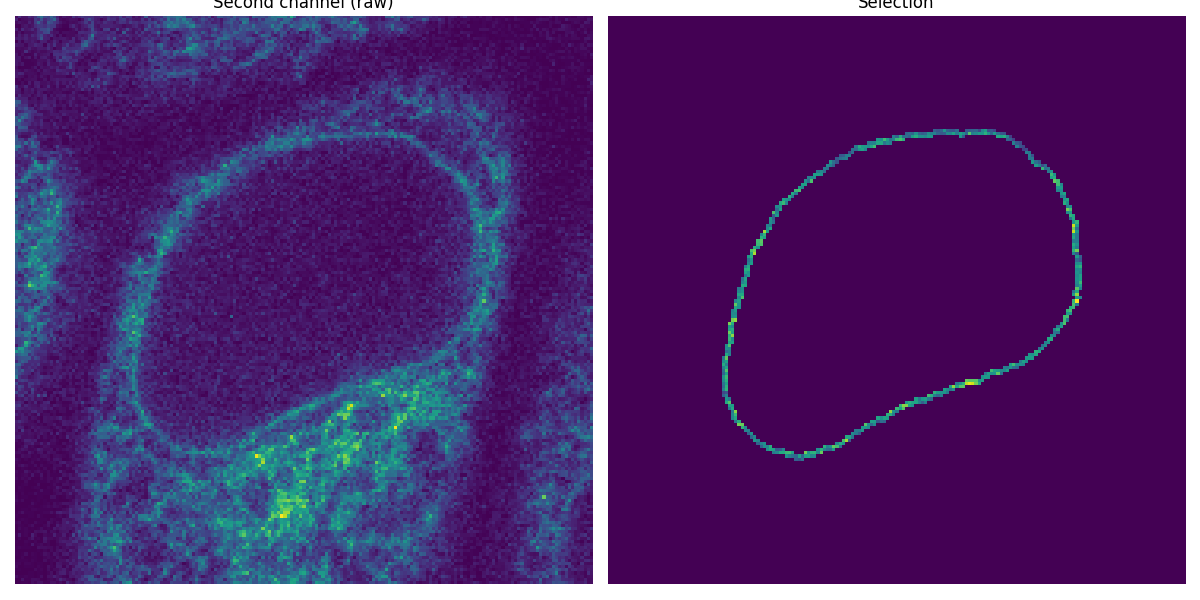
セグメント化された縁をマスクとして適用#
最初のチャネルで核膜をセグメント化したので、それをマスクとして使用して、2番目のチャネルの強度を測定します。
image_t_0_channel_1 = image_sequence[0, 1, :, :]
selection = np.where(mask, image_t_0_channel_1, 0)
fig, (ax0, ax1) = plt.subplots(1, 2, figsize=(12, 6), sharey=True)
ax0.imshow(image_t_0_channel_1)
ax0.set_title('Second channel (raw)')
ax0.set_axis_off()
ax1.imshow(selection)
ax1.set_title('Selection')
ax1.set_axis_off()
fig.tight_layout()