注記
最後に移動して、完全なサンプルコードをダウンロードするか、Binder経由でブラウザでこの例を実行してください
3D画像(細胞)の探索#
このチュートリアルは、3次元画像処理の入門です。3Dデータセットの簡単な紹介については、3つ以上の空間次元を持つデータセットを参照してください。画像はnumpy配列として表現されます。単一チャネルまたはグレースケール画像は、形状(n_row, n_col)のピクセル強度を持つ2D行列です。ここで、n_row(resp. n_col)はrows(resp. columns)の数を表します。一連の2Dplanesとして3Dボリュームを構築でき、3D画像に形状(n_plane, n_row, n_col)を与えます。ここで、n_planeはプレーンの数です。マルチチャネルまたはRGB(A)画像には、色情報を含む最後の位置に追加のchannel次元があります。
これらの規則を以下の表にまとめます。
画像タイプ |
座標 |
|---|---|
2Dグレースケール |
|
2Dマルチチャネル |
|
3Dグレースケール |
|
3Dマルチチャネル |
|
一部の3D画像は、各次元で等しい解像度で構築されます(例:シンクロトロン断層撮影または球のコンピューター生成レンダリング)。しかし、ほとんどの実験データは、3次元のうちの1つで低い解像度でキャプチャされます。たとえば、薄いスライスを撮影して、2D画像のスタックとして3D構造を近似します。各次元のピクセル間の距離(間隔と呼ばれる)はタプルとしてエンコードされ、一部のskimage関数でパラメーターとして受け入れられ、フィルターへの寄与を調整するために使用できます。
このチュートリアルで使用されているデータは、Allen Institute for Cell Scienceから提供されました。サイズを縮小し、計算時間を短縮するために、rowおよびcolumn次元で4分の1にダウンサンプリングされました。間隔情報は、細胞の画像化に使用された顕微鏡によって報告されました。
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d.art3d import Poly3DCollection
import numpy as np
import plotly
import plotly.express as px
from skimage import exposure, util
from skimage.data import cells3d
3D画像のロードと表示#
data = util.img_as_float(cells3d()[:, 1, :, :]) # grab just the nuclei
print(f'shape: {data.shape}')
print(f'dtype: {data.dtype}')
print(f'range: ({data.min()}, {data.max()})')
# Report spacing from microscope
original_spacing = np.array([0.2900000, 0.0650000, 0.0650000])
# Account for downsampling of slices by 4
rescaled_spacing = original_spacing * [1, 4, 4]
# Normalize spacing so that pixels are a distance of 1 apart
spacing = rescaled_spacing / rescaled_spacing[2]
print(f'microscope spacing: {original_spacing}\n')
print(f'rescaled spacing: {rescaled_spacing} (after downsampling)\n')
print(f'normalized spacing: {spacing}\n')
shape: (60, 256, 256)
dtype: float64
range: (0.0, 1.0)
microscope spacing: [0.29 0.065 0.065]
rescaled spacing: [0.29 0.26 0.26] (after downsampling)
normalized spacing: [1.11538462 1. 1. ]
3D画像を視覚化してみましょう。残念ながら、matplotlibのimshowなどの多くの画像ビューアーは、2Dデータのみを表示できます。3Dデータを表示しようとすると、エラーが発生することがわかります。
Invalid shape (60, 256, 256) for image data
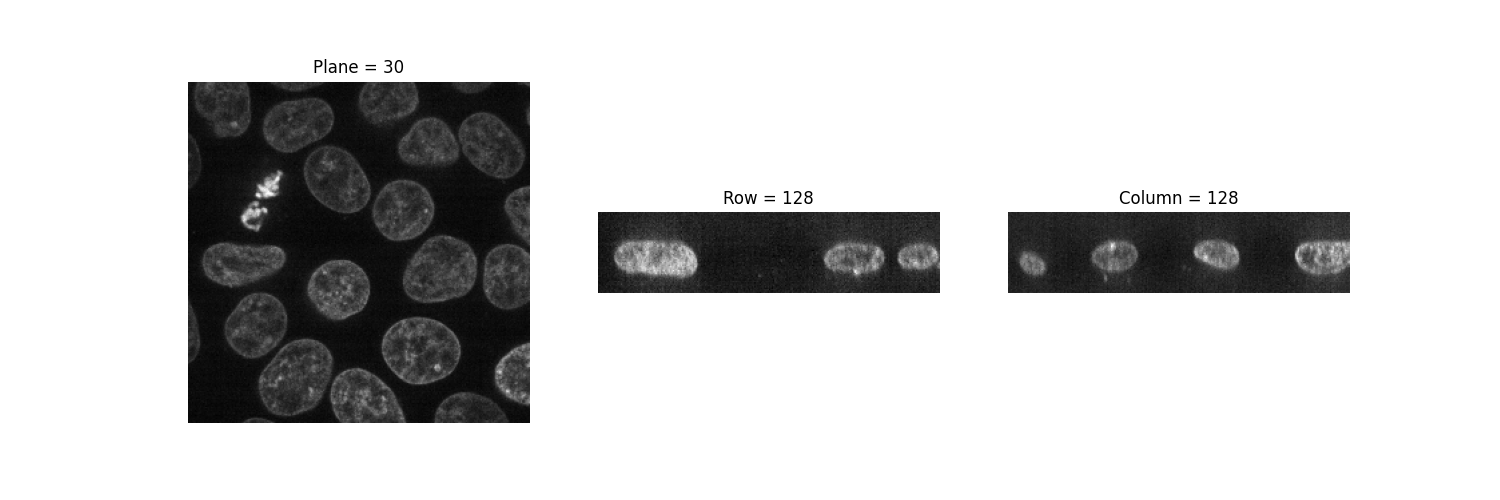
imshow関数は、グレースケールおよびRGB(A)2D画像のみを表示できます。したがって、それを使用して2D平面を視覚化できます。1つの軸を固定することにより、画像の3つの異なるビューを観察できます。
def show_plane(ax, plane, cmap="gray", title=None):
ax.imshow(plane, cmap=cmap)
ax.set_axis_off()
if title:
ax.set_title(title)
(n_plane, n_row, n_col) = data.shape
_, (a, b, c) = plt.subplots(ncols=3, figsize=(15, 5))
show_plane(a, data[n_plane // 2], title=f'Plane = {n_plane // 2}')
show_plane(b, data[:, n_row // 2, :], title=f'Row = {n_row // 2}')
show_plane(c, data[:, :, n_col // 2], title=f'Column = {n_col // 2}')
前に示したように、3次元画像は一連の二次元平面として見ることができます。いくつかの平面のモンタージュを作成するヘルパー関数displayを記述してみましょう。デフォルトでは、他のすべての平面が表示されます。
def display(im3d, cmap='gray', step=2):
data_montage = util.montage(im3d[::step], padding_width=4, fill=np.nan)
_, ax = plt.subplots(figsize=(16, 14))
ax.imshow(data_montage, cmap=cmap)
ax.set_axis_off()
display(data)

または、Jupyterウィジェットを使用してこれらの平面(スライス)をインタラクティブに探索できます。ユーザーに表示するスライスを選択させ、3Dデータセットでのこのスライスの位置を示します。この例のオンライン版のように、静的なHTMLページでは、Jupyterウィジェットが動作しているのを確認することはできません。次のコードが機能するためには、ローカルまたはクラウドで実行されているJupyterカーネルが必要です。このページの下部を参照して、Jupyterノートブックをダウンロードしてコンピューターで実行するか、Binderで直接開いてください。アクティブなカーネルに加えて、Webブラウザが必要です。純粋なPythonでコードを実行しても機能しません。
def slice_in_3D(ax, i):
# From https://stackoverflow.com/questions/44881885/python-draw-3d-cube
Z = np.array(
[
[0, 0, 0],
[1, 0, 0],
[1, 1, 0],
[0, 1, 0],
[0, 0, 1],
[1, 0, 1],
[1, 1, 1],
[0, 1, 1],
]
)
Z = Z * data.shape
r = [-1, 1]
X, Y = np.meshgrid(r, r)
# Plot vertices
ax.scatter3D(Z[:, 0], Z[:, 1], Z[:, 2])
# List sides' polygons of figure
verts = [
[Z[0], Z[1], Z[2], Z[3]],
[Z[4], Z[5], Z[6], Z[7]],
[Z[0], Z[1], Z[5], Z[4]],
[Z[2], Z[3], Z[7], Z[6]],
[Z[1], Z[2], Z[6], Z[5]],
[Z[4], Z[7], Z[3], Z[0]],
[Z[2], Z[3], Z[7], Z[6]],
]
# Plot sides
ax.add_collection3d(
Poly3DCollection(
verts, facecolors=(0, 1, 1, 0.25), linewidths=1, edgecolors="darkblue"
)
)
verts = np.array([[[0, 0, 0], [0, 0, 1], [0, 1, 1], [0, 1, 0]]])
verts = verts * (60, 256, 256)
verts += [i, 0, 0]
ax.add_collection3d(
Poly3DCollection(verts, facecolors="magenta", linewidths=1, edgecolors="black")
)
ax.set_xlabel("plane")
ax.set_xlim(0, 100)
ax.set_ylabel("row")
ax.set_zlabel("col")
# Autoscale plot axes
scaling = np.array([getattr(ax, f'get_{dim}lim')() for dim in "xyz"])
ax.auto_scale_xyz(*[[np.min(scaling), np.max(scaling)]] * 3)
def explore_slices(data, cmap="gray"):
from ipywidgets import interact
N = len(data)
@interact(plane=(0, N - 1))
def display_slice(plane=34):
fig, ax = plt.subplots(figsize=(20, 5))
ax_3D = fig.add_subplot(133, projection="3d")
show_plane(ax, data[plane], title=f'Plane {plane}', cmap=cmap)
slice_in_3D(ax_3D, plane)
plt.show()
return display_slice
explore_slices(data)
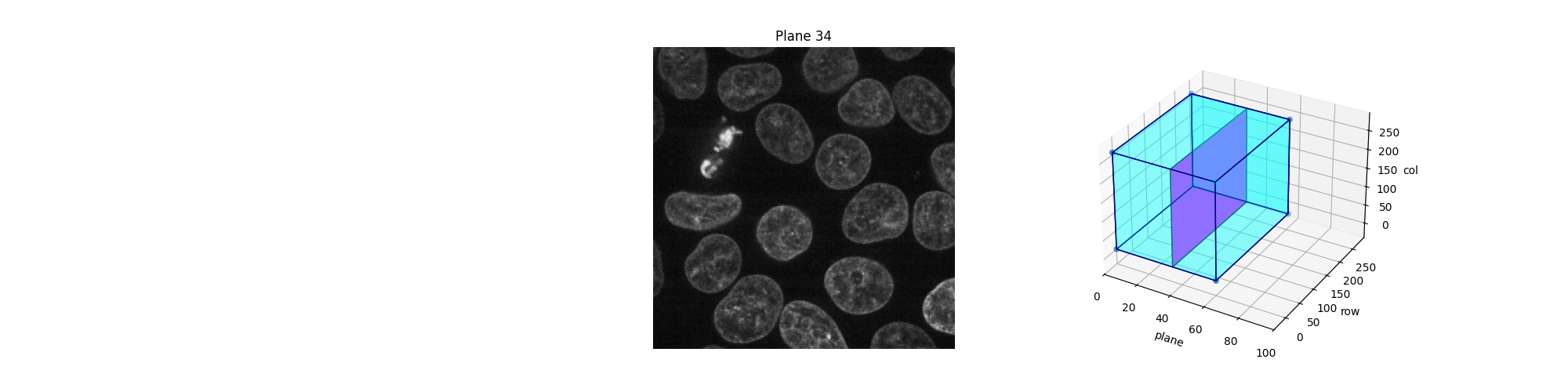
interactive(children=(IntSlider(value=34, description='plane', max=59), Output()), _dom_classes=('widget-interact',))
<function explore_slices.<locals>.display_slice at 0x7f287ebaa020>
露出の調整#
scikit-imageのexposureモジュールには、画像のコントラストを調整するための多くの関数が含まれています。これらの関数は、ピクセル値に対して動作します。一般に、画像次元またはピクセル間隔を考慮する必要はありません。ただし、局所露出補正では、各軸に沿って実際の座標でサイズが等しくなるように、ウィンドウサイズを調整する必要がある場合があります。
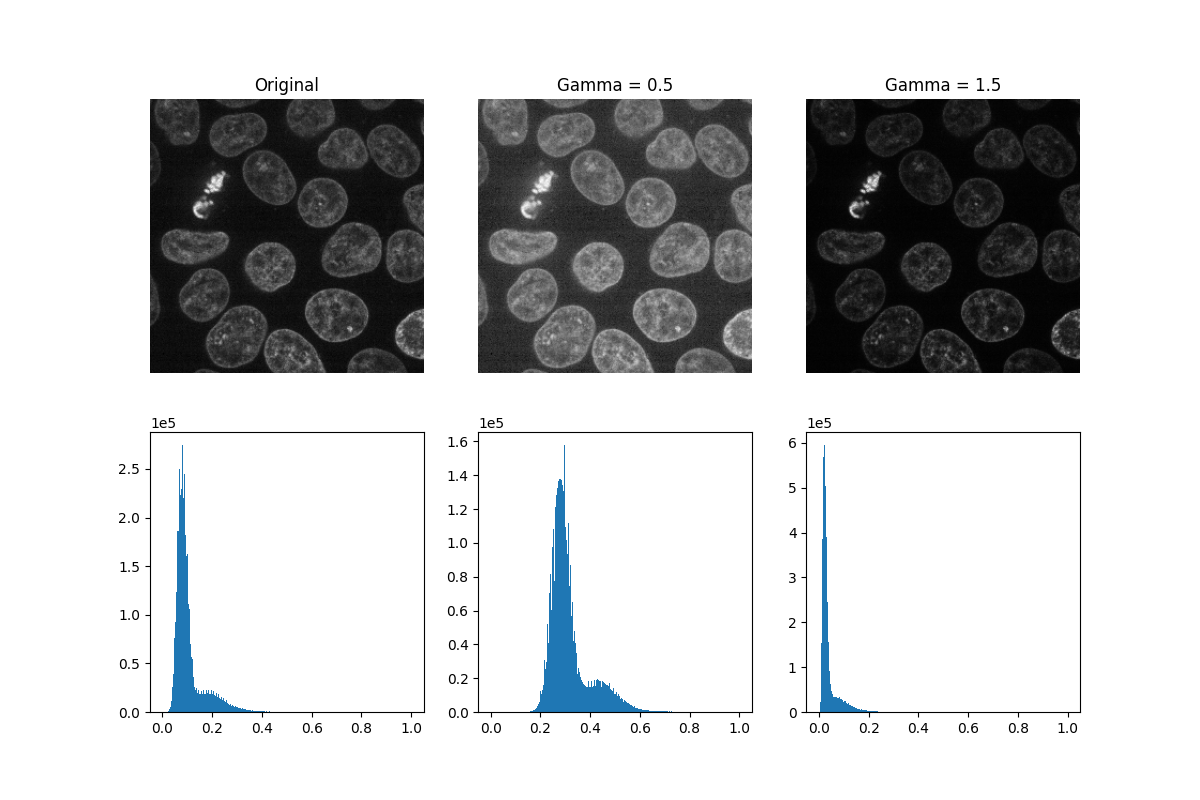
ガンマ補正は、画像を明るくまたは暗くします。べき乗則変換(ここで、gammaはべき乗則指数を表します)が画像の各ピクセルに適用されます。gamma < 1は画像を明るくし、gamma > 1は画像を暗くします。
def plot_hist(ax, data, title=None):
# Helper function for plotting histograms
ax.hist(data.ravel(), bins=256)
ax.ticklabel_format(axis="y", style="scientific", scilimits=(0, 0))
if title:
ax.set_title(title)
gamma_low_val = 0.5
gamma_low = exposure.adjust_gamma(data, gamma=gamma_low_val)
gamma_high_val = 1.5
gamma_high = exposure.adjust_gamma(data, gamma=gamma_high_val)
_, ((a, b, c), (d, e, f)) = plt.subplots(nrows=2, ncols=3, figsize=(12, 8))
show_plane(a, data[32], title='Original')
show_plane(b, gamma_low[32], title=f'Gamma = {gamma_low_val}')
show_plane(c, gamma_high[32], title=f'Gamma = {gamma_high_val}')
plot_hist(d, data)
plot_hist(e, gamma_low)
plot_hist(f, gamma_high)
ヒストグラム平坦化は、ピクセル強度を再配分することで画像のコントラストを改善します。最も一般的なピクセル強度は分散され、コントラストの低い領域のコントラストを向上させます。このアプローチの欠点の1つは、背景ノイズを強調してしまう可能性があることです。
equalized_data = exposure.equalize_hist(data)
display(equalized_data)

これまでと同様に、Jupyterカーネルが実行されていれば、上記のスライスをインタラクティブに探索できます。
explore_slices(equalized_data)
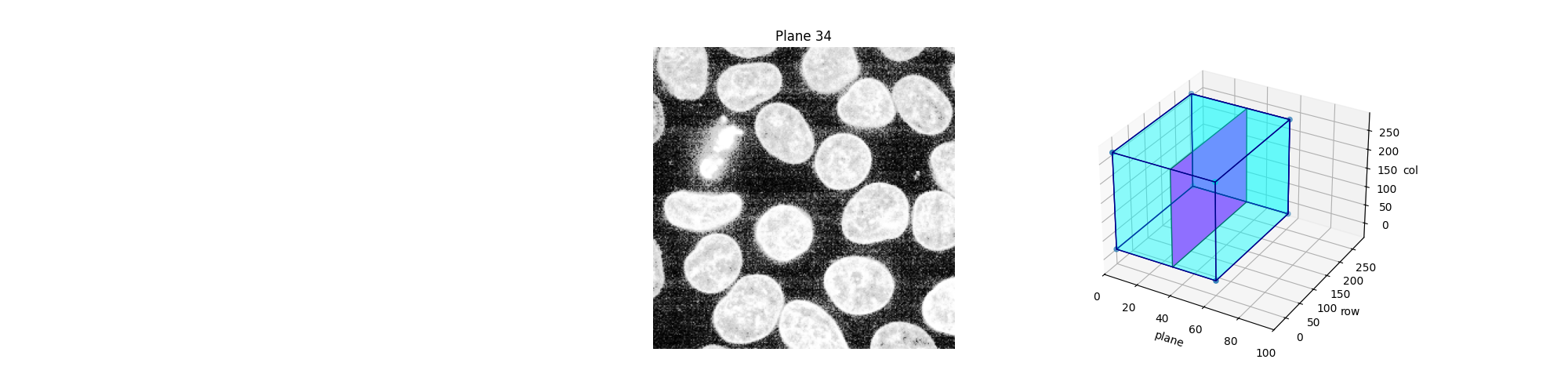
interactive(children=(IntSlider(value=34, description='plane', max=59), Output()), _dom_classes=('widget-interact',))
<function explore_slices.<locals>.display_slice at 0x7f287efc22a0>
次に、ヒストグラム平坦化の前後の画像のヒストグラムをプロットしてみましょう。以下に、それぞれの累積分布関数(CDF)をプロットします。
_, ((a, b), (c, d)) = plt.subplots(nrows=2, ncols=2, figsize=(16, 8))
plot_hist(a, data, title="Original histogram")
plot_hist(b, equalized_data, title="Equalized histogram")
cdf, bins = exposure.cumulative_distribution(data.ravel())
c.plot(bins, cdf, "r")
c.set_title("Original CDF")
cdf, bins = exposure.cumulative_distribution(equalized_data.ravel())
d.plot(bins, cdf, "r")
d.set_title("Histogram equalization CDF")
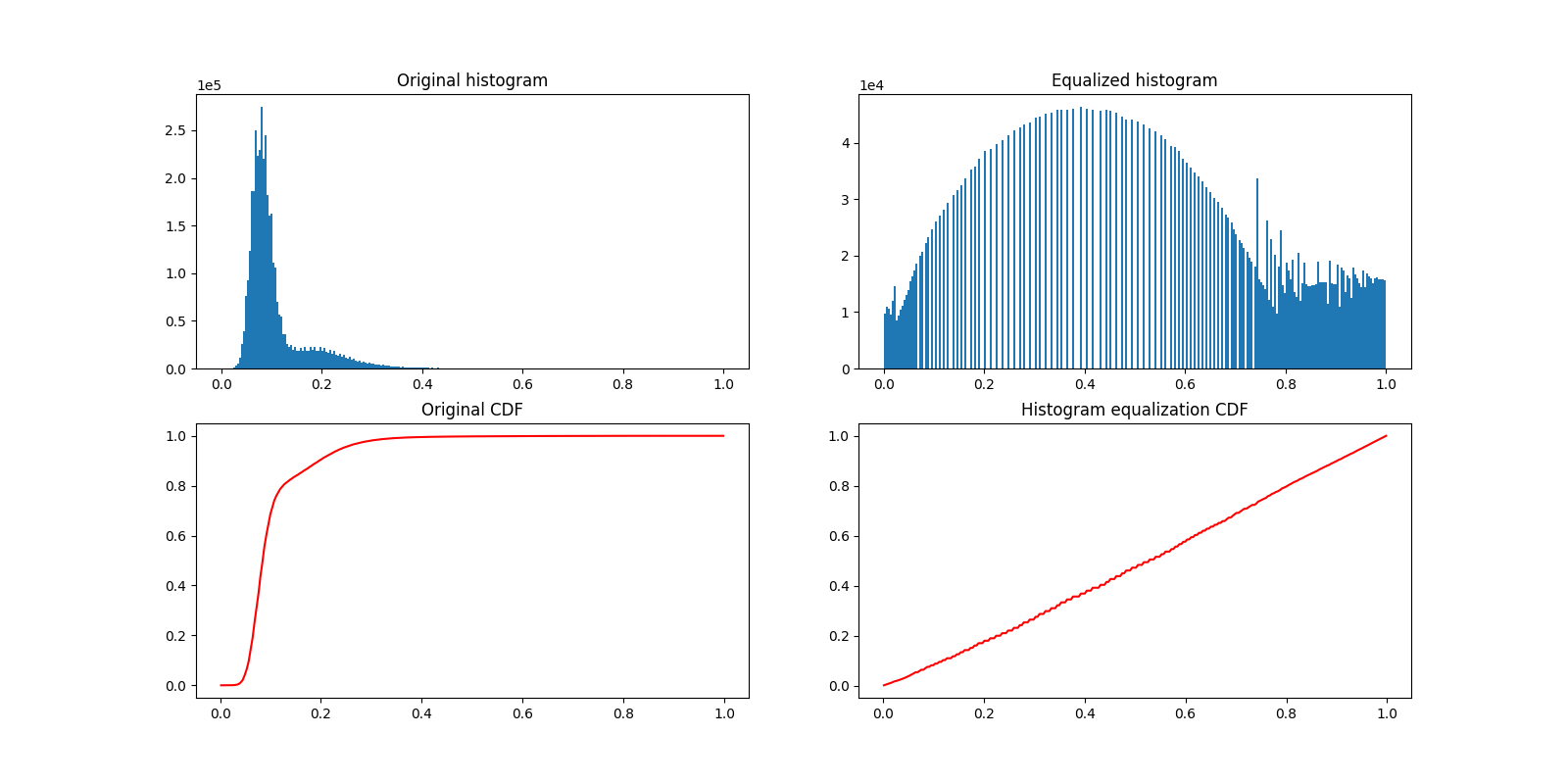
Text(0.5, 1.0, 'Histogram equalization CDF')
ほとんどの実験画像は、塩コショウノイズの影響を受けます。少数の明るいアーチファクトは、関心のあるピクセルの相対強度を低下させる可能性があります。コントラストを改善する簡単な方法として、ピクセル値を最低値と最高値の両極端でクリップすることが挙げられます。最も暗いピクセルと最も明るいピクセルの0.5%をクリップすると、画像の全体的なコントラストが向上します。
vmin, vmax = np.percentile(data, q=(0.5, 99.5))
clipped_data = exposure.rescale_intensity(
data, in_range=(vmin, vmax), out_range=np.float32
)
display(clipped_data)

あるいは、Plotly Expressを使用して、これらの平面(スライス)をインタラクティブに探索することもできます。これは静的なHTMLページでも機能することに注意してください!
fig = px.imshow(data, animation_frame=0, binary_string=True)
fig.update_xaxes(showticklabels=False)
fig.update_yaxes(showticklabels=False)
fig.update_layout(autosize=False, width=500, height=500, coloraxis_showscale=False)
# Drop animation buttons
fig['layout'].pop('updatemenus')
plotly.io.show(fig)
plt.show()
スクリプトの総実行時間: (0分10.688秒)
