skimage.exposure#
画像強度調整(例:ヒストグラム平坦化など)
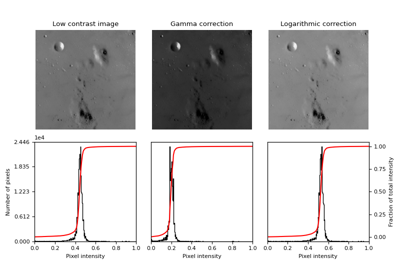
入力画像に対してガンマ補正を実行します。 |
|
入力画像に対して対数補正を実行します。 |
|
入力画像に対してシグモイド補正を実行します。 |
|
指定された画像の累積分布関数(cdf)を返します。 |
|
コントラスト制限適応ヒストグラム平坦化(CLAHE)。 |
|
ヒストグラム平坦化後の画像を返します。 |
|
画像のヒストグラムを返します。 |
|
画像が低コントラストかどうかを判断します。 |
|
累積ヒストグラムが別の画像のヒストグラムと一致するように画像を調整します。 |
|
強度レベルを拡大または縮小した後、画像を返します。 |
- skimage.exposure.adjust_gamma(image, gamma=1, gain=1)[ソース]#
入力画像に対してガンマ補正を実行します。
パワー法則変換としても知られています。この関数は、各ピクセルを0〜1の範囲にスケーリングした後、方程式
O = I**gammaに従って入力画像をピクセル単位で変換します。- パラメータ:
- imagendarray
入力画像。
- gammafloat、オプション
非負の実数。デフォルト値は1です。
- gainfloat、オプション
定数乗数。デフォルト値は1です。
- 戻り値:
- outndarray
ガンマ補正された出力画像。
参考
注釈
ガンマが1より大きい場合、ヒストグラムは左にシフトし、出力画像は入力画像よりも暗くなります。
ガンマが1より小さい場合、ヒストグラムは右にシフトし、出力画像は入力画像よりも明るくなります。
参考文献
例
>>> from skimage import data, exposure, img_as_float >>> image = img_as_float(data.moon()) >>> gamma_corrected = exposure.adjust_gamma(image, 2) >>> # Output is darker for gamma > 1 >>> image.mean() > gamma_corrected.mean() True
- skimage.exposure.adjust_log(image, gain=1, inv=False)[ソース]#
入力画像に対して対数補正を実行します。
この関数は、各ピクセルを0〜1の範囲にスケーリングした後、方程式
O = gain*log(1 + I)に従って入力画像をピクセル単位で変換します。逆対数補正の場合、方程式はO = gain*(2**I - 1)です。- パラメータ:
- imagendarray
入力画像。
- gainfloat、オプション
定数乗数。デフォルト値は1です。
- invfloat、オプション
Trueの場合、逆対数補正を実行し、それ以外の場合は補正は対数になります。デフォルトはFalseです。
- 戻り値:
- outndarray
対数補正された出力画像。
参考
参考文献
- skimage.exposure.adjust_sigmoid(image, cutoff=0.5, gain=10, inv=False)[ソース]#
入力画像に対してシグモイド補正を実行します。
コントラスト調整としても知られています。この関数は、各ピクセルを0〜1の範囲にスケーリングした後、方程式
O = 1/(1 + exp*(gain*(cutoff - I)))に従って入力画像をピクセル単位で変換します。- パラメータ:
- imagendarray
入力画像。
- cutofffloat、オプション
シグモイド関数のカットオフ。特徴曲線を水平方向にシフトします。デフォルト値は0.5です。
- gainfloat、オプション
シグモイド関数の指数部に使用される定数乗数。デフォルト値は10です。
- invbool、オプション
Trueの場合、負のシグモイド補正を返します。デフォルトはFalseです。
- 戻り値:
- outndarray
シグモイド補正された出力画像。
参考
参考文献
[1]Gustav J. Braun, “Image Lightness Rescaling Using Sigmoidal Contrast Enhancement Functions”, http://markfairchild.org/PDFs/PAP07.pdf
- skimage.exposure.cumulative_distribution(image, nbins=256)[ソース]#
指定された画像の累積分布関数(cdf)を返します。
- パラメータ:
- image配列
画像配列。
- nbinsint、オプション
画像ヒストグラムのビン数。
- 戻り値:
- img_cdf配列
累積分布関数の値。
- bin_centers配列
ビンの中心。
参考
参考文献
例
>>> from skimage import data, exposure, img_as_float >>> image = img_as_float(data.camera()) >>> hi = exposure.histogram(image) >>> cdf = exposure.cumulative_distribution(image) >>> all(cdf[0] == np.cumsum(hi[0])/float(image.size)) True
- skimage.exposure.equalize_adapthist(image, kernel_size=None, clip_limit=0.01, nbins=256)[ソース]#
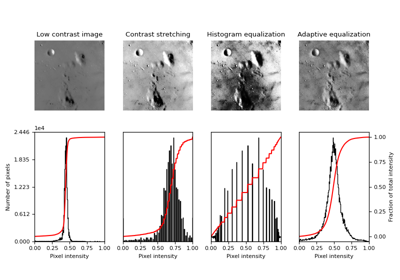
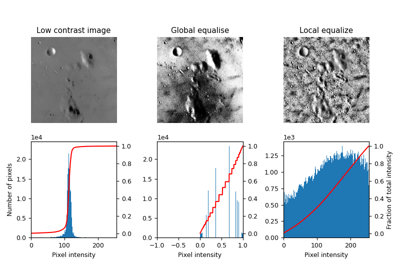
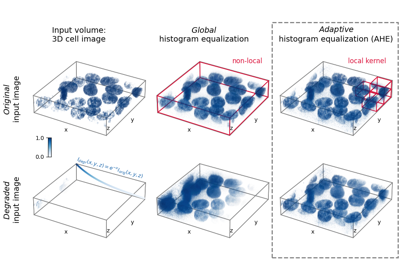
コントラスト制限適応ヒストグラム平坦化(CLAHE)。
画像の異なるタイル領域で計算されたヒストグラムを使用する、局所コントラスト強調のアルゴリズム。したがって、局所的な詳細は、画像の大部分よりも暗いまたは明るい領域でも強調できます。
- パラメータ:
- image(M[, …][, C]) ndarray
入力画像。
- kernel_sizeintまたはarray_like、オプション
アルゴリズムで使用される文脈領域の形状を定義します。iterableが渡された場合、
image.ndim(カラーチャネルなし)と同じ数の要素を持つ必要があります。整数の場合、各image次元にブロードキャストされます。デフォルトでは、kernel_sizeは、imageの高さの1/8、幅の1/8です。- clip_limitfloat、オプション
クリッピング制限。0〜1の間で正規化されます(値が大きいほどコントラストが強くなります)。
- nbinsint、オプション
ヒストグラムのグレイビン数(「データ範囲」)。
- 戻り値:
- out(M[, …][, C]) ndarray
float64 dtypeを使用した平坦化された画像。
注釈
- カラー画像の場合、次の手順が実行されます。
画像はHSV色空間に変換されます
CLAHEアルゴリズムはV(値)チャネルで実行されます
画像はRGB空間に戻され、返されます
RGBA画像の場合、元のアルファチャネルは削除されます。
バージョン0.17で変更: この関数によって返される値は、丸め動作の内部的な変更により、わずかに上にシフトしています。
参考文献
- skimage.exposure.equalize_hist(image, nbins=256, mask=None)[ソース]#
ヒストグラム平坦化後の画像を返します。
- パラメータ:
- image配列
画像配列。
- nbinsint、オプション
画像ヒストグラムのビン数。注:この引数は、各整数が独自のビンである整数画像では無視されます。
- maskboolまたは0と1のndarray、オプション
imageと同じ形状の配列。マスク== Trueのポイントのみが平坦化に使用され、画像全体に適用されます。
- 戻り値:
- outfloat配列
ヒストグラム平坦化後の画像配列。
注釈
この関数は、著者の許可を得て[1]から採用されました。
参考文献
- skimage.exposure.histogram(image, nbins=256, source_range='image', normalize=False, *, channel_axis=None)[source]#
画像のヒストグラムを返します。
numpy.histogramとは異なり、この関数はビンの中心を返し、整数配列をリビンしません。整数配列の場合、各整数値は独自のビンを持ち、速度と強度分解能が向上します。channel_axisが設定されていない場合、ヒストグラムは平坦化された画像に対して計算されます。カラー画像またはマルチチャネル画像の場合、channel_axisを設定して、すべてのチャネルで共通のビニングを使用します。または、各チャネルで関数を個別に適用して、個別のビニングを持つ各カラーチャネルのヒストグラムを取得することもできます。- パラメータ:
- image配列
入力画像。
- nbinsint、オプション
ヒストグラムの計算に使用するビンの数。この値は整数配列では無視されます。
- source_rangestring, オプション
「image」(デフォルト)は、入力画像から範囲を決定します。「dtype」は、そのデータ型の画像の予想される範囲から範囲を決定します。
- normalizebool, オプション
Trueの場合、ヒストグラムをその値の合計で正規化します。
- channel_axisint または None, オプション
Noneの場合、画像はグレースケール(シングルチャネル)画像と見なされます。それ以外の場合、このパラメーターは、配列のどの軸がチャネルに対応するかを示します。
- 戻り値:
- histarray
ヒストグラムの値。
channel_axisが None でない場合、hist は最初の軸がチャネルに対応する2次元配列になります。- bin_centers配列
ビンの中心の値。
例
>>> from skimage import data, exposure, img_as_float >>> image = img_as_float(data.camera()) >>> np.histogram(image, bins=2) (array([ 93585, 168559]), array([0. , 0.5, 1. ])) >>> exposure.histogram(image, nbins=2) (array([ 93585, 168559]), array([0.25, 0.75]))
- skimage.exposure.is_low_contrast(image, fraction_threshold=0.05, lower_percentile=1, upper_percentile=99, method='linear')[source]#
画像が低コントラストかどうかを判断します。
- パラメータ:
- imagearray-like
テスト対象の画像。
- fraction_thresholdfloat, オプション
低コントラストの閾値となる割合。画像の明るさの範囲が、データ型の全範囲のこの割合よりも小さい場合、画像は低コントラストと見なされます。[1]
- lower_percentilefloat, オプション
画像のコントラストを計算するときに、このパーセンタイルを下回る値を無視します。
- upper_percentilefloat, オプション
画像のコントラストを計算するときに、このパーセンタイルを上回る値を無視します。
- methodstr, オプション
コントラストの決定方法。現在利用可能なオプションは「linear」のみです。
- 戻り値:
- outbool
画像が低コントラストと判定された場合は True。
注釈
ブール画像の場合、すべての値が同じ場合にのみこの関数は False を返します(method、threshold、および percentile 引数は無視されます)。
参考文献
例
>>> image = np.linspace(0, 0.04, 100) >>> is_low_contrast(image) True >>> image[-1] = 1 >>> is_low_contrast(image) True >>> is_low_contrast(image, upper_percentile=100) False
- skimage.exposure.match_histograms(image, reference, *, channel_axis=None)[source]#
累積ヒストグラムが別の画像のヒストグラムと一致するように画像を調整します。
調整は、チャネルごとに個別に適用されます。
- パラメータ:
- imagendarray
入力画像。グレースケールまたはカラーにすることができます。
- referencendarray
ヒストグラムを一致させる画像。画像と同じ数のチャネルが必要です。
- channel_axisint または None, オプション
Noneの場合、画像はグレースケール(シングルチャネル)画像と見なされます。それ以外の場合、このパラメーターは、配列のどの軸がチャネルに対応するかを示します。
- 戻り値:
- matchedndarray
変換された入力画像。
- Raises:
- ValueError
入力画像と参照のチャネル数が異なる場合にスローされます。
参考文献
- skimage.exposure.rescale_intensity(image, in_range='image', out_range='dtype')[source]#
強度レベルを拡大または縮小した後、画像を返します。
入力と出力の目的の強度範囲である
in_rangeとout_rangeを使用して、入力画像の強度範囲を拡大または縮小します。以下の例を参照してください。- パラメータ:
- image配列
画像配列。
- in_range, out_rangestr または 2-tuple, オプション
入力画像と出力画像の最小および最大強度値。このパラメーターで使用できる値は、以下に列挙されています。
- 「image」
画像の最小/最大値を強度範囲として使用します。
- 「dtype」
画像の dtype の最小/最大値を強度範囲として使用します。
- dtype-name
目的の
dtypeに基づいた強度範囲を使用します。DTYPE_RANGEの有効なキーである必要があります。- 2-tuple
range_valuesを明示的な最小/最大強度として使用します。
- 戻り値:
- outarray
強度を再スケーリングした後の画像配列。この画像は、入力画像と同じ dtype です。
注釈
バージョン 0.17 で変更: 出力配列の dtype は、入力の dtype と一致するように変更されました。または、出力範囲が値のペアで指定されている場合は float になります。
例
デフォルトでは、
in_rangeが「image」に、out_rangeが「dtype」にデフォルト設定されているため、入力画像の最小/最大強度は、画像の dtype で許可される範囲の限界まで拡大されます。>>> image = np.array([51, 102, 153], dtype=np.uint8) >>> rescale_intensity(image) array([ 0, 127, 255], dtype=uint8)
画像 dtype を uint8 から float に誤って変換するのは簡単です
>>> 1.0 * image array([ 51., 102., 153.])
rescale_intensityを使用して、float dtype の適切な範囲に再スケールします>>> image_float = 1.0 * image >>> rescale_intensity(image_float) array([0. , 0.5, 1. ])
元の低コントラストを維持するには、
in_rangeパラメーターを使用します>>> rescale_intensity(image_float, in_range=(0, 255)) array([0.2, 0.4, 0.6])
in_rangeの最小/最大値が最小/最大画像強度よりも大きい/小さい場合、強度レベルはクリップされます>>> rescale_intensity(image_float, in_range=(0, 102)) array([0.5, 1. , 1. ])
符号付き整数を持つ画像があるが、画像を正の範囲のみに再スケールする場合は、
out_rangeパラメーターを使用します。この場合、出力 dtype は float になります>>> image = np.array([-10, 0, 10], dtype=np.int8) >>> rescale_intensity(image, out_range=(0, 127)) array([ 0. , 63.5, 127. ])
特定の dtype で目的の範囲を取得するには、
.astype()を使用します>>> rescale_intensity(image, out_range=(0, 127)).astype(np.int8) array([ 0, 63, 127], dtype=int8)
入力画像が一定の場合、出力は出力範囲に直接クリップされます。>>> image = np.array([130, 130, 130], dtype=np.int32) >>> rescale_intensity(image, out_range=(0, 127)).astype(np.int32) array([127, 127, 127], dtype=int32)