注記
最後まで移動して、完全なサンプルコードをダウンロードします。または、Binder経由でブラウザでこの例を実行します。
pandas を使用した領域特性の探索と可視化#
この簡単な例では、10枚の画像シリーズの各ラベル付き領域のサイズを計算する方法を示します。2D画像と3D画像を使用します。ブロブ状の領域は合成的に生成されます。体積分率(つまり、ブロブによって覆われたピクセルまたはボクセルの比率)が増加すると、ブロブ(領域)の数は減少し、単一領域のサイズ(面積または体積)は大きくなります。面積(サイズ)の値はpandasと互換性のある形式で利用できるため、便利なデータ分析と可視化が可能になります。
面積以外にも、多くの領域特性が利用可能です。
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import seaborn as sns
from skimage import data, measure
fractions = np.linspace(0.05, 0.5, 10)
2D画像#
images = [data.binary_blobs(volume_fraction=f) for f in fractions]
labeled_images = [measure.label(image) for image in images]
properties = ['label', 'area']
tables = [
measure.regionprops_table(image, properties=properties) for image in labeled_images
]
tables = [pd.DataFrame(table) for table in tables]
for fraction, table in zip(fractions, tables):
table['volume fraction'] = fraction
areas = pd.concat(tables, axis=0)
# Create custom grid of subplots
grid = plt.GridSpec(2, 2)
ax1 = plt.subplot(grid[0, 0])
ax2 = plt.subplot(grid[0, 1])
ax = plt.subplot(grid[1, :])
# Show image with lowest volume fraction
ax1.imshow(images[0], cmap='gray_r')
ax1.set_axis_off()
ax1.set_title(f'fraction {fractions[0]}')
# Show image with highest volume fraction
ax2.imshow(images[-1], cmap='gray_r')
ax2.set_axis_off()
ax2.set_title(f'fraction {fractions[-1]}')
# Plot area vs volume fraction
areas.plot(x='volume fraction', y='area', kind='scatter', ax=ax)
plt.show()
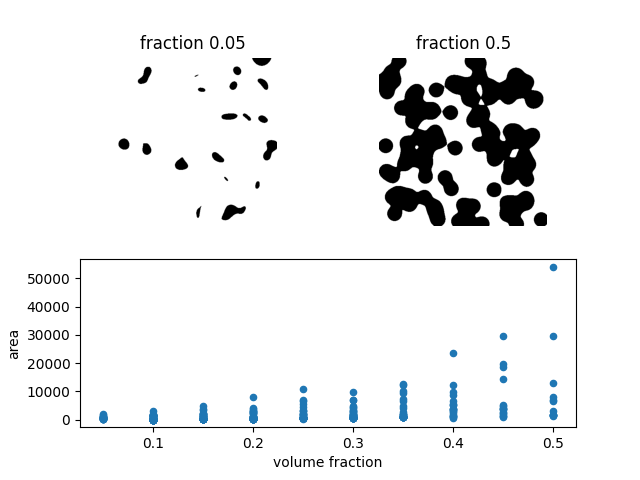
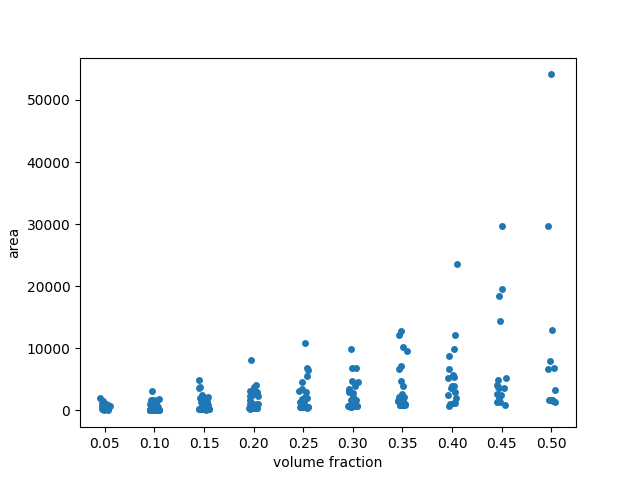
散布図では、多くの点が小さな面積値で重なっているように見えます。分布をよりよく理解するために、可視化にいくつかの「ジッター」を追加したい場合があります。この目的のために、引数jitter=Trueを使用してseaborn.stripplot(統計データ可視化のためのseabornライブラリから)を使用します。
fig, ax = plt.subplots()
sns.stripplot(x='volume fraction', y='area', data=areas, jitter=True, ax=ax)
# Fix floating point rendering
ax.set_xticklabels([f'{frac:.2f}' for frac in fractions])
plt.show()
/home/runner/work/scikit-image/scikit-image/doc/examples/segmentation/plot_regionprops_table.py:75: UserWarning:
set_ticklabels() should only be used with a fixed number of ticks, i.e. after set_ticks() or using a FixedLocator.
3D画像#
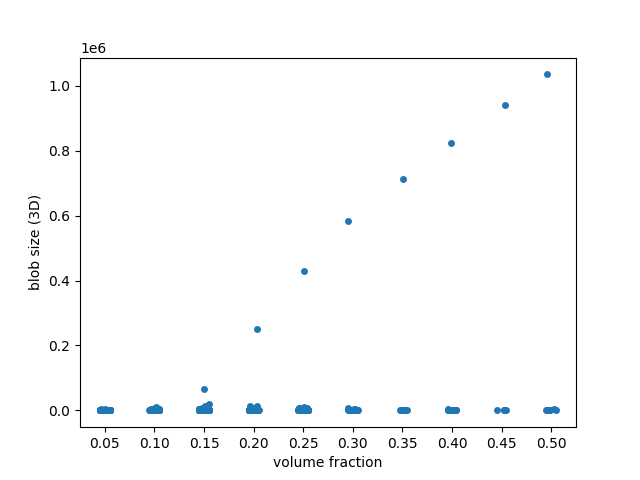
3Dで同じ分析を行うと、はるかに劇的な挙動が見られます。体積分率が約0.25を超えると、ブロブは単一の巨大な塊に合体します。これは、統計物理学とグラフ理論におけるパーコレーション閾値に対応します。
images = [data.binary_blobs(length=128, n_dim=3, volume_fraction=f) for f in fractions]
labeled_images = [measure.label(image) for image in images]
properties = ['label', 'area']
tables = [
measure.regionprops_table(image, properties=properties) for image in labeled_images
]
tables = [pd.DataFrame(table) for table in tables]
for fraction, table in zip(fractions, tables):
table['volume fraction'] = fraction
blob_volumes = pd.concat(tables, axis=0)
fig, ax = plt.subplots()
sns.stripplot(x='volume fraction', y='area', data=blob_volumes, jitter=True, ax=ax)
ax.set_ylabel('blob size (3D)')
# Fix floating point rendering
ax.set_xticklabels([f'{frac:.2f}' for frac in fractions])
plt.show()
/home/runner/work/scikit-image/scikit-image/doc/examples/segmentation/plot_regionprops_table.py:107: UserWarning:
set_ticklabels() should only be used with a fixed number of ticks, i.e. after set_ticks() or using a FixedLocator.
スクリプトの総実行時間:(0分4.787秒)
