4. NumPy の画像のクイックコース#
scikit-image の画像は NumPy ndarray で表されます。したがって、多くの一般的な操作は、配列を操作するための標準 NumPy メソッドを使用して実現できます
>>> import skimage as ski
>>> camera = ski.data.camera()
>>> type(camera)
<type 'numpy.ndarray'>
注意
pandas.DataFrame や xarray.DataArray などのラベル付き配列のようなデータタイプは scikit-image でもともとサポートされていません。ただし、これらのタイプに格納されたデータは、numpy.ndarray に変換できます(pandas.DataFrame.to_numpy() と xarray.DataArray.data を参照)。特に、これらの変換はサンプリング座標(DataFrame.index、DataFrame.columns、または DataArray.coords)を無視します。これは、本来のデータポイントが不規則に間隔を置かれている場合などに、誤って表現されたデータになる可能性があります。
画像のジオメトリとピクセル数の取得
>>> camera.shape
(512, 512)
>>> camera.size
262144
画像の輝度値に関する統計情報の取得
>>> camera.min(), camera.max()
(0, 255)
>>> camera.mean()
118.31400299072266
画像を表す NumPy 配列は、異なる整数または浮動小数点のタイプの数値にすることができます。これらのタイプと scikit-imageがそれらを処理する方法の詳細については、イメージデータのタイプとそれらの意味を参照してください。
4.1. NumPy インデックス#
NumPy のインデックスは、ピクセル値の表示と変更の両方に使用できます
>>> # Get the value of the pixel at the 10th row and 20th column
>>> camera[10, 20]
153
>>> # Set to black the pixel at the 3rd row and 10th column
>>> camera[3, 10] = 0
NumPy インデックス付けでは最初の次元 (camera.shape[0]) が行、2番目の次元 (camera.shape[1]) が列に相当し、原点が (camera[0, 0]) 左上隅になります。これは行列/線形代数表記に合いますが、デカルト (x, y) 座標とは対照的です。詳細は下にある 座標の規約 を参照してください。
個別ピクセルを越えて、NumPy のさまざまなインデックス付け機能を使用して、ピクセル全体のセットの値にアクセス/変更することが可能です。
スライス
>>> # Set the first ten lines to "black" (0)
>>> camera[:10] = 0
マスク処理 (ブール値のマスクによるインデックス付け)
>>> mask = camera < 87
>>> # Set to "white" (255) the pixels where mask is True
>>> camera[mask] = 255
ファンシーインデックス付け (インデックスのセットによるインデックス付け)
>>> import numpy as np
>>> inds_r = np.arange(len(camera))
>>> inds_c = 4 * inds_r % len(camera)
>>> camera[inds_r, inds_c] = 0
マスクは処理を実行するピクセルセットを選択する必要がある場合に非常に役立ちます。マスクは画像と同じ形状 (または画像形状にブロードキャスト可能な形状) のブール配列にできます。これは興味領域 (円など) を定義するために使用できます。
>>> nrows, ncols = camera.shape
>>> row, col = np.ogrid[:nrows, :ncols]
>>> cnt_row, cnt_col = nrows / 2, ncols / 2
>>> outer_disk_mask = ((row - cnt_row)**2 + (col - cnt_col)**2 >
... (nrows / 2)**2)
>>> camera[outer_disk_mask] = 0

NumPy のブール演算を使用して、さらに複雑なマスクも定義できます。
>>> lower_half = row > cnt_row
>>> lower_half_disk = np.logical_and(lower_half, outer_disk_mask)
>>> camera = data.camera()
>>> camera[lower_half_disk] = 0
4.2. カラー画像#
上記のすべてはカラー画像にも当てはまります。カラー画像はチャネル用の追加の末尾次元を持つ NumPy 配列です。
>>> cat = ski.data.chelsea()
>>> type(cat)
<type 'numpy.ndarray'>
>>> cat.shape
(300, 451, 3)
これは cat が 3 つのチャネル (赤、緑、青) を持つ 300 x 451 ピクセルの画像であることを示しています。これまでと同様に、ピクセル値を取得して設定できます。
>>> cat[10, 20]
array([151, 129, 115], dtype=uint8)
>>> # Set the pixel at (50th row, 60th column) to "black"
>>> cat[50, 60] = 0
>>> # set the pixel at (50th row, 61st column) to "green"
>>> cat[50, 61] = [0, 255, 0] # [red, green, blue]
上記のグレースケール画像で行ったように、2 次元マルチチャネル画像にも 2 次元のブールマスクを使用できます。
(ソース コード, png, hires.png, pdf)
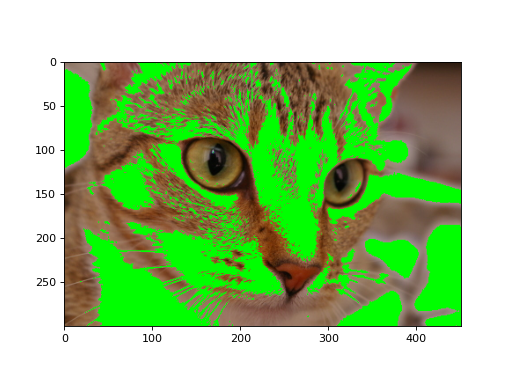
2 次元カラー画像に 2 次元マスクを使用する#
skimage.data に含まれるサンプルのカラー画像は最後の軸にチャネルが格納されていますが、他のソフトウェアでは異なる規約に従う場合があります。カラー画像をサポートする scikit-image ライブラリ関数は channel_axis 引数を持っています。この引数は配列のどの軸がチャネルに対応するかを指定するために使用できます。
4.3. 座標の規約#
scikit-image が NumPy 配列を使用して画像を表現するため、座標の規則をそれに合わせてなければなりません。2 次元(2D)のグレースケール画像(上記の camera など)は行と列によってインデックス付けされ((row, col) または (r, c) に省略)、左上の角にある最小の要素 (0, 0) があります。ライブラリのさまざまな部分では、rr と cc が行と列の座標のリストを参照しているのを見かけます。この規則は、標準的なデカルト座標を表す (x, y) とは区別されます。x は水平座標で、y は垂直座標で、原点は左下にあります(例えば、Matplotlib の軸はこの規則を使用しています)。
多チャンネル画像の場合、いずれの次元(配列軸)もカラーチャンネルに使用できます。これは channel または ch で表されます。scikit-image 0.19 より前では、このチャンネル次元は常に最後でしたが、現在のリリースではチャンネル次元は channel_axis 引数で指定できます。多チャンネルデータが必要な関数は、デフォルトで channel_axis=-1 になります。それ以外の場合は、関数のデフォルトは channel_axis=None になり、 هیچ محورもチャンネルに対応していないことを示します。
最後に、ビデオ、磁気共鳴画像(MRI)スキャン、共焦点顕微鏡などの画像などのボリューム(3D)画像の場合、先行次元を plane と呼び、pln または p と省略します。
これらの規則を以下にまとめます。
イメージの種類 |
座標 |
|---|---|
2D グレースケール |
(行、列) |
2D 多チャンネル(例:RGB) |
(行、列、チャンネル) |
3D グレースケール |
(平面、行、列) |
3D 多チャンネル |
(平面、行、列、チャンネル) |
chの位置は、channel_axis 引数によって制御されることに注意してください。
scikit-image の多くの関数は、3D 画像で直接操作できます。
>>> import numpy as np
>>> import scipy as sp
>>> import skimage as ski
>>> rng = np.random.default_rng()
>>> im3d = rng.random((100, 1000, 1000))
>>> seeds = sp.ndimage.label(im3d < 0.1)[0]
>>> ws = ski.segmentation.watershed(im3d, seeds)
ただし、多くの場合、3 つ目の空間次元の解像度は他の 2 つより低くなります。scikit-image 関数のいくつかには、この種のデータを処理するのに役立つ spacing キーワード引数が提供されています。
>>> slics = ski.segmentation.slic(im3d, spacing=[5, 1, 1], channel_axis=None)
他の場合、処理は面ごとに実行する必要があります。面が先行次元(規約に従って)に沿って積み重ねられている場合、次の構文を使用できます。
>>> edges = np.empty_like(im3d)
>>> for pln, image in enumerate(im3d):
... # Iterate over the leading dimension
... edges[pln] = ski.filters.sobel(image)
4.4 配列の次元の順序に関する注意#
軸のラベル付けは恣意的に見えるかもしれませんが、演算の速度に重大な影響を与える可能性があります。これは、最新のプロセッサはメモリから 1 つの項目だけを取得するのではなく、近隣の項目のチャンク全体(プリフェッチと呼ばれる操作)を取得するためです。したがって、メモリ内で隣接する要素の処理は、操作の数は同じ場合でも、それらが散在している場合に処理するよりも高速です。
>>> def in_order_multiply(arr, scalar):
... for plane in list(range(arr.shape[0])):
... arr[plane, :, :] *= scalar
...
>>> def out_of_order_multiply(arr, scalar):
... for plane in list(range(arr.shape[2])):
... arr[:, :, plane] *= scalar
...
>>> import time
>>> rng = np.random.default_rng()
>>> im3d = rng.random((100, 1024, 1024))
>>> t0 = time.time(); x = in_order_multiply(im3d, 5); t1 = time.time()
>>> print("%.2f seconds" % (t1 - t0))
0.14 seconds
>>> s0 = time.time(); x = out_of_order_multiply(im3d, 5); s1 = time.time()
>>> print("%.2f seconds" % (s1 - s0))
1.18 seconds
>>> print("Speedup: %.1fx" % ((s1 - s0) / (t1 - t0)))
Speedup: 8.6x
最後/右端の次元がさらに大きくなると、スピードアップはさらに劇的になります。アルゴリズムを開発する際にデータの局所性について考える価値があります。特に、scikit-imageはデフォルトで C に隣接する配列を使用します。ネストされたループを使用する場合、配列の最後/右端の次元は、計算の最も内側のループ内にあります。上記の例では、*= numpy 演算子はすべての残りの次元を反復処理します。
4.5 時間次元の注意事項#
scikit-imageは現在、時間変化する 3D データで具体的に動作する関数を提供していませんが、NumPy 配列との互換性により、(t、pln、row、col、ch) の形状を持つ 5D 配列を非常に自然に動作させることができます。
>>> for timepoint in image5d:
... # Each timepoint is a 3D multichannel image
... do_something_with(timepoint)
そこで、上記の表を次のように補完することができます。
イメージの種類 |
座標 |
|---|---|
2D カラー ビデオ |
(t、行、列、ch) |
3D カラー ビデオ |
(t、pln、行、列、ch) |
