注記
完全なサンプルコードをダウンロードするには、最後まで進むか、Binder経由でブラウザでこの例を実行してください。
インペイントによる斑点のある角膜画像の復元#
光干渉断層計(OCT)は、眼科医が患者の眼の奥の写真を撮るために使用する非侵襲的な画像技術です [1]。OCTを実施する際に、機器の参照ミラーに埃が付着して、画像に黒い斑点が現れることがあります。問題は、これらの汚れの斑点がin vivo組織の領域を覆い、関心のあるデータを隠してしまうことです。ここでの目標は、境界付近のピクセルに基づいて、隠れた領域を復元(再構築)することです。
このチュートリアルは、Jules Scholler [2]が共有したアプリケーションを基にしています。画像はViacheslav Mazlinによって取得されました(skimage.data.palisades_of_vogt()を参照)。
import matplotlib.pyplot as plt
import numpy as np
import plotly.io
import plotly.express as px
import skimage as ski
ここで使用しているデータセットは、ヒトin vivo組織の画像シーケンス(動画!)です。具体的には、特定の角膜サンプルの*Vogt柵状体*を示しています。
画像データの読み込み#
image_seq = ski.data.palisades_of_vogt()
print(f'number of dimensions: {image_seq.ndim}')
print(f'shape: {image_seq.shape}')
print(f'dtype: {image_seq.dtype}')
number of dimensions: 3
shape: (60, 1440, 1440)
dtype: uint16
データセットは、60フレーム(時点)と2つの空間次元を持つ画像スタックです。6時点ごとにサンプリングすることで10フレームを可視化してみましょう。照度の変化が見られます。Plotlyの`imshow`関数の`animation_frame`パラメータを利用しています。ちなみに、`binary_string`パラメータが`True`に設定されている場合、画像はグレースケールで表示されます。
fig = px.imshow(
image_seq[::6, :, :],
animation_frame=0,
binary_string=True,
labels={'animation_frame': '6-step time point'},
title='Sample of in-vivo human cornea',
)
fig.update_layout(autosize=False, minreducedwidth=250, minreducedheight=250)
plotly.io.show(fig)
時間をかけて集計する#
まず、データが失われた汚れの斑点を検出したいと考えています。専門用語では、汚れの斑点(シーケンスのすべてのフレーム)を*セグメント化*したいということです。実際のデータ(信号)とは異なり、汚れの斑点はフレーム間で移動しません。静止しています。そのため、まず画像シーケンスの時間集計を計算することから始めます。汚れの斑点をセグメント化するために中央値画像を使用します。汚れの斑点は背景(ぼやけた信号)に対して目立つようになります。補完的に、(移動する)データの感触をつかむために、分散を計算してみましょう。
image_med = np.median(image_seq, axis=0)
image_var = np.var(image_seq, axis=0)
assert image_var.shape == image_med.shape
print(f'shape: {image_med.shape}')
fig, ax = plt.subplots(ncols=2, figsize=(12, 6))
ax[0].imshow(image_med, cmap='gray')
ax[0].set_title('Image median over time')
ax[1].imshow(image_var, cmap='gray')
ax[1].set_title('Image variance over time')
fig.tight_layout()

shape: (1440, 1440)
局所しきい値処理を使用する#
汚れの斑点をセグメント化するために、しきい値処理を使用します。操作対象の画像は照度が不均一であるため、前景と背景の(絶対)強度に空間的なばらつきが生じます。たとえば、ある領域の平均背景強度が、別の(遠く離れた)領域とは異なる場合があります。そのため、画像全体で異なるしきい値を、各領域ごとに1つずつ計算する方が適切です。これは、画像内のすべてのピクセルに単一の(グローバル)しきい値を使用する通常のしきい値処理手順とは対照的に、適応(または局所)しきい値処理と呼ばれます。
`filters`モジュールの`threshold_local`関数を呼び出す際には、デフォルトの近傍サイズ(`block_size`)、つまり照度が変化する典型的なサイズ(ピクセル数)と、`offset`(近傍の加重平均をシフトする)を変更できます。`block_size`に2つの異なる値を試してみましょう。
2つのプロットを並べて表示する便利な関数を定義して、比較しやすくしましょう。
def plot_comparison(plot1, plot2, title1, title2):
fig, (ax1, ax2) = plt.subplots(ncols=2, figsize=(12, 6), sharex=True, sharey=True)
ax1.imshow(plot1, cmap='gray')
ax1.set_title(title1)
ax2.imshow(plot2, cmap='gray')
ax2.set_title(title2)
plot_comparison(mask_1, mask_2, "block_size = 21", "block_size = 43")

「汚れの斑点」は2番目のマスク、つまりより大きな`block_size`値を使用した結果得られたマスクの方がはっきりと見えるようです。オフセットパラメータの値をデフォルトのゼロ値から増やすと、背景がより均一になり、関心のあるオブジェクトがより目立つようになることがわかりました。パラメータ値を切り替えることで、使用されているメソッドをより深く理解できるようになり、通常は目的の結果に近づけることができます。

きめ細かい特徴を除去する#
マスクを鮮明化し、汚れの斑点に焦点を当てるために、モルフォロジーフィルターを使用します。2つの基本的なモルフォロジー演算子は、*膨張*と*収縮*です。膨張(収縮)は、構造化要素(フットプリント)によって定義された近傍の最も明るい(暗い)値にピクセルを設定します。
ここでは、`morphology`モジュールの`diamond`関数を使用して、ダイヤモンド型のフットプリントを作成します。収縮に続いて膨張を行うことを*オープニング*と呼びます。まず、オープニングフィルターを適用して、小さなオブジェクトと細い線を取り除きながら、大きなオブジェクトの形状とサイズを維持します。
footprint = ski.morphology.diamond(3)
mask_open = ski.morphology.opening(mask_2, footprint)
plot_comparison(mask_2, mask_open, "mask before", "after opening")

画像の「オープニング」は収縮操作から始まるため、構造化要素よりも小さい明るい領域は削除されます。オープニングフィルターを適用する場合、`footprint` パラメータを調整することで、削除される特徴の細かさを制御できます。たとえば、上記の例で `footprint = ski.morphology.diamond(1)` を使用した場合、より小さな特徴のみがフィルタリングされるため、マスク内のスポットがより多く保持されます。逆に、同じ半径の円盤状のフットプリント、つまり `footprint = ski.morphology.disk(3)` を使用した場合、円盤の面積はダイヤモンドよりも大きいため、より細かい特徴がフィルタリングされます。
次に、膨張フィルターを適用することで、検出された領域を広くすることができます。
mask_dilate = ski.morphology.dilation(mask_open, footprint)
plot_comparison(mask_open, mask_dilate, "Before", "After dilation")
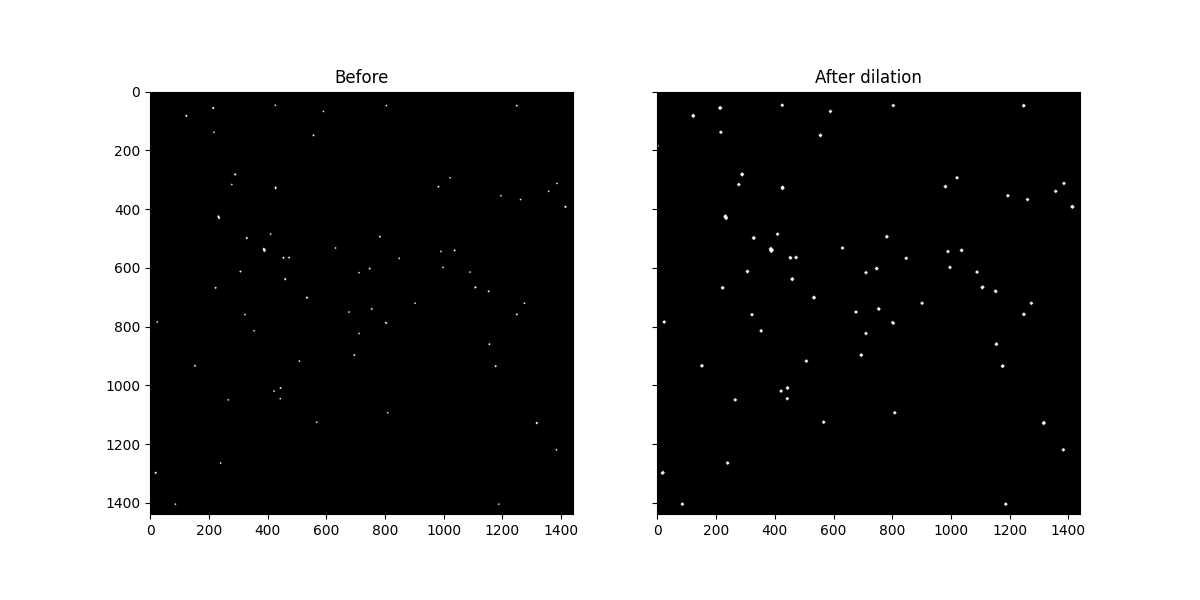
膨張は明るい領域を拡大し、暗い領域を縮小します。 実際に、白い斑点がどのように厚くなっているかに注目してください。
各フレームを個別に修復する#
これで、各フレームに修復を適用する準備が整いました。このために、`restoration` モジュールから関数 `inpaint_biharmonic` を使用します。これは、重調和方程式に基づくアルゴリズムを実装しています。この関数は、2つの配列を入力として受け取ります。修復する画像と、修復したい領域に対応するマスク(同じ形状)です。
image_seq_inpainted = np.zeros(image_seq.shape)
for i in range(image_seq.shape[0]):
image_seq_inpainted[i] = ski.restoration.inpaint_biharmonic(
image_seq[i], mask_dilate
)
汚れの斑点が修復された、復元された画像の1つを視覚化してみましょう。 まず、汚れの斑点(つまり、マスク)の輪郭を見つけ、復元された画像の上に描画できるようにします。
contours = ski.measure.find_contours(mask_dilate)
# Gather all (row, column) coordinates of the contours
x = []
y = []
for contour in contours:
x.append(contour[:, 0])
y.append(contour[:, 1])
# Note that the following one-liner is equivalent to the above:
# x, y = zip(*((contour[:, 0], contour[:, 1]) for contour in contours))
# Flatten the coordinates
x_flat = np.concatenate(x).ravel().round().astype(int)
y_flat = np.concatenate(y).ravel().round().astype(int)
# Create mask of these contours
contour_mask = np.zeros(mask_dilate.shape, dtype=bool)
contour_mask[x_flat, y_flat] = 1
# Pick one frame
sample_result = image_seq_inpainted[12]
# Normalize it (so intensity values range [0, 1])
sample_result /= sample_result.max()
`color` モジュールから関数 `label2rgb` を使用して、透明度(アルファパラメータ)を使用して、復元された画像にセグメント化されたスポットを重ね合わせます。
color_contours = ski.color.label2rgb(
contour_mask, image=sample_result, alpha=0.4, bg_color=(1, 1, 1)
)
fig, ax = plt.subplots(figsize=(6, 6))
ax.imshow(color_contours)
ax.set_title('Segmented spots over restored image')
fig.tight_layout()
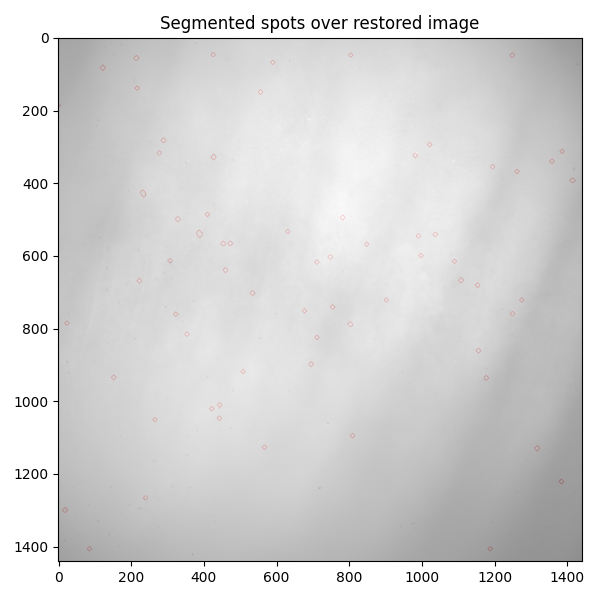
(x, y) ~ (719, 1237) にある汚れの斑点が際立っていることに注意してください。理想的には、セグメント化されて修復されるべきでした。細かい特徴を削除するオープニング処理ステップで、それを「失った」ことがわかります。
plt.show()
**スクリプトの合計実行時間:** (0分29.329秒)
