skimage.filters.rank#
局所ヒストグラムを用いた画像の自動レベル調整。 |
|
画像のグレースケール局所自動レベル調整を返します。 |
|
画像のコントラストを強調します。 |
|
画像のコントラストを強調します。 |
|
局所エントロピー。 |
|
局所ヒストグラムを用いた画像のヒストグラム均一化。 |
|
画像の局所幾何平均を返します。 |
|
画像の局所勾配(つまり、局所最大値 - 局所最小値)を返します。 |
|
画像の局所勾配(つまり、局所最大値 - 局所最小値)を返します。 |
|
各ピクセルに、その近傍領域内で最も一般的な値を割り当てます。 |
|
画像の局所最大値を返します。 |
|
画像の局所平均を返します。 |
|
フラットカーネル双方向フィルタを適用します。 |
|
画像の局所平均を返します。 |
|
画像の局所中央値を返します。 |
|
画像の局所最小値を返します。 |
|
画像の局所モードを返します。 |
|
ノイズ特徴量。 |
|
各ピクセルに対する局所的な大津の閾値。 |
|
画像の局所パーセンタイルを返します。 |
|
局所的なピクセル数(個体数)を返します。 |
|
局所的なピクセル数(個体数)を返します。 |
|
局所的なピクセル数(個体数)を返します。 |
|
局所平均から引いた画像を返します。 |
|
局所平均から引いた画像を返します。 |
|
ピクセルの局所合計を返します。 |
|
フラットカーネル双方向フィルタを適用します。 |
|
ピクセルの局所合計を返します。 |
|
画像の局所閾値。 |
|
画像の局所閾値。 |
|
正規化されたスライディングウィンドウヒストグラム |
- skimage.filters.rank.autolevel(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
局所ヒストグラムを用いた画像の自動レベル調整。
このフィルタは、グレースケール値のヒストグラムを局所的に伸張し、「白」から「黒」までの値の全範囲をカバーします。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
例
>>> from skimage import data >>> from skimage.morphology import disk, ball >>> from skimage.filters.rank import autolevel >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> auto = autolevel(img, disk(5)) >>> auto_vol = autolevel(volume, ball(5))
- skimage.filters.rank.autolevel_percentile(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, p0=0, p1=1)[source]#
画像のグレースケール局所自動レベル調整を返します。
このフィルタは、グレースケール値のヒストグラムを局所的に伸張し、「白」から「黒」までの値の全範囲をカバーします。
パーセンタイル[p0, p1]の間のグレースケール値のみがフィルタで考慮されます。
- パラメータ:
- image2次元配列 (uint8, uint16)
入力画像。
- footprint2次元配列
1と0の2次元配列で表された近傍領域。
- out2次元配列 (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- p0, p1[0, …, 1] の範囲内の浮動小数点数
値の計算に使用する[p0, p1]パーセンタイル間隔を定義します。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
- skimage.filters.rank.enhance_contrast(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
画像のコントラストを強調します。
これは、ピクセルのグレースケール値が局所最小値よりも局所最大値に近い場合、各ピクセルを局所最大値に置き換えます。そうでない場合は、局所最小値に置き換えられます。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像
例
>>> from skimage import data >>> from skimage.morphology import disk, ball >>> from skimage.filters.rank import enhance_contrast >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> out = enhance_contrast(img, disk(5)) >>> out_vol = enhance_contrast(volume, ball(5))
- skimage.filters.rank.enhance_contrast_percentile(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, p0=0, p1=1)[source]#
画像のコントラストを強調します。
これは、ピクセルのグレースケール値が局所最小値よりも局所最大値に近い場合、各ピクセルを局所最大値に置き換えます。そうでない場合は、局所最小値に置き換えられます。
パーセンタイル[p0, p1]の間のグレースケール値のみがフィルタで考慮されます。
- パラメータ:
- image2次元配列 (uint8, uint16)
入力画像。
- footprint2次元配列
1と0の2次元配列で表された近傍領域。
- out2次元配列 (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- p0, p1[0, …, 1] の範囲内の浮動小数点数
値の計算に使用する[p0, p1]パーセンタイル間隔を定義します。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
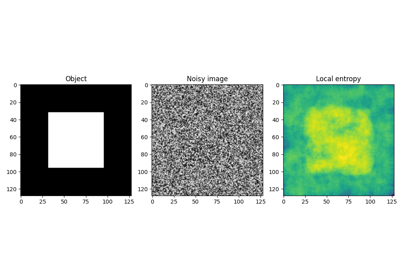
- skimage.filters.rank.entropy(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
局所エントロピー。
エントロピーは、底を2とする対数を使用して計算されます。つまり、このフィルタは、局所的なグレースケールレベル分布をエンコードするために必要な最小ビット数を返します。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (float)
出力画像。
参考文献
例
>>> from skimage import data >>> from skimage.filters.rank import entropy >>> from skimage.morphology import disk, ball >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> ent = entropy(img, disk(5)) >>> ent_vol = entropy(volume, ball(5))
- skimage.filters.rank.equalize(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
局所ヒストグラムを用いた画像のヒストグラム均一化。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
例
>>> from skimage import data >>> from skimage.morphology import disk, ball >>> from skimage.filters.rank import equalize >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> equ = equalize(img, disk(5)) >>> equ_vol = equalize(volume, ball(5))
- skimage.filters.rank.geometric_mean(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
画像の局所幾何平均を返します。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
参考文献
[1]Gonzalez, R. C. and Woods, R. E. “Digital Image Processing (3rd Edition).” Prentice-Hall Inc, 2006.
例
>>> from skimage import data >>> from skimage.morphology import disk, ball >>> from skimage.filters.rank import mean >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> avg = geometric_mean(img, disk(5)) >>> avg_vol = geometric_mean(volume, ball(5))
- skimage.filters.rank.gradient(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
画像の局所勾配(つまり、局所最大値 - 局所最小値)を返します。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
例
>>> from skimage import data >>> from skimage.morphology import disk, ball >>> from skimage.filters.rank import gradient >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> out = gradient(img, disk(5)) >>> out_vol = gradient(volume, ball(5))
- skimage.filters.rank.gradient_percentile(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, p0=0, p1=1)[source]#
画像の局所勾配(つまり、局所最大値 - 局所最小値)を返します。
パーセンタイル[p0, p1]の間のグレースケール値のみがフィルタで考慮されます。
- パラメータ:
- image2次元配列 (uint8, uint16)
入力画像。
- footprint2次元配列
1と0の2次元配列で表された近傍領域。
- out2次元配列 (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- p0, p1[0, …, 1] の範囲内の浮動小数点数
値の計算に使用する[p0, p1]パーセンタイル間隔を定義します。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
- skimage.filters.rank.majority(image, footprint, *, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
各ピクセルに、その近傍領域内で最も一般的な値を割り当てます。
- パラメータ:
- imagendarray
画像配列(uint8、uint16配列)。
- footprint2次元配列(整数または浮動小数点数)
1と0の2次元配列で表された近傍領域。
- outndarray(整数または浮動小数点数)、オプション
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint、オプション
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
例
>>> from skimage import data >>> from skimage.filters.rank import majority >>> from skimage.morphology import disk, ball >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> maj_img = majority(img, disk(5)) >>> maj_img_vol = majority(volume, ball(5))
- skimage.filters.rank.maximum(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
画像の局所最大値を返します。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
備考
アルゴリズムの計算量が小さいことから、
skimage.filters.rank.maximumは、より大きな画像とフットプリントに対してより効率的です。例
>>> from skimage import data >>> from skimage.morphology import disk, ball >>> from skimage.filters.rank import maximum >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> out = maximum(img, disk(5)) >>> out_vol = maximum(volume, ball(5))
- skimage.filters.rank.mean(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
画像の局所平均を返します。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
例
>>> from skimage import data >>> from skimage.morphology import disk, ball >>> from skimage.filters.rank import mean >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> avg = mean(img, disk(5)) >>> avg_vol = mean(volume, ball(5))
- skimage.filters.rank.mean_bilateral(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, s0=10, s1=10)[source]#
フラットカーネル双方向フィルタを適用します。
これは、エッジを保持し、ノイズを低減するノイズ除去フィルターです。画素の空間的な近接性と放射輝度の類似性に基づいて、画素の平均化を行います。
空間的な近接性は、フットプリント(構造要素)によって与えられる局所的な画素近傍のみを考慮することで測定されます。
放射輝度の類似性は、gが現在の画素のグレイレベルである場合、グレイレベル区間[g-s0, g+s1]によって定義されます。
フットプリントに属し、この区間内にグレイレベルを持つ画素のみが平均化されます。
- パラメータ:
- image2次元配列 (uint8, uint16)
入力画像。
- footprint2次元配列
1と0の2次元配列で表された近傍領域。
- out2次元配列 (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- s0, s1int
値の計算に考慮される中心画素のグレイ値を中心とした[s0, s1]区間を定義します。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
例
>>> import numpy as np >>> from skimage import data >>> from skimage.morphology import disk >>> from skimage.filters.rank import mean_bilateral >>> img = data.camera().astype(np.uint16) >>> bilat_img = mean_bilateral(img, disk(20), s0=10,s1=10)
- skimage.filters.rank.mean_percentile(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, p0=0, p1=1)[source]#
画像の局所平均を返します。
パーセンタイル[p0, p1]の間のグレースケール値のみがフィルタで考慮されます。
- パラメータ:
- image2次元配列 (uint8, uint16)
入力画像。
- footprint2次元配列
1と0の2次元配列で表された近傍領域。
- out2次元配列 (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- p0, p1[0, …, 1] の範囲内の浮動小数点数
値の計算に使用する[p0, p1]パーセンタイル間隔を定義します。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
- skimage.filters.rank.median(image, footprint=None, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
画像の局所中央値を返します。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
近傍領域は、1と0のndarrayで表現されます。Noneの場合、サイズは3の完全な正方形が使用されます。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
参照
skimage.filters.median浮動小数点精度を持つ画像を処理するメディアンフィルタリングの実装です。
例
>>> from skimage import data >>> from skimage.morphology import disk, ball >>> from skimage.filters.rank import median >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> med = median(img, disk(5)) >>> med_vol = median(volume, ball(5))
- skimage.filters.rank.minimum(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
画像の局所最小値を返します。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
備考
アルゴリズムの計算量が低いことから、
skimage.filters.rank.minimumは、より大きな画像とフットプリントに対してより効率的です。例
>>> from skimage import data >>> from skimage.morphology import disk, ball >>> from skimage.filters.rank import minimum >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> out = minimum(img, disk(5)) >>> out_vol = minimum(volume, ball(5))
- skimage.filters.rank.modal(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
画像の局所モードを返します。
モードとは、局所ヒストグラムで最も頻繁に現れる値です。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
例
>>> from skimage import data >>> from skimage.morphology import disk, ball >>> from skimage.filters.rank import modal >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> out = modal(img, disk(5)) >>> out_vol = modal(volume, ball(5))
- skimage.filters.rank.noise_filter(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
ノイズ特徴量。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
参考文献
[1]N. Hashimoto et al. Referenceless image quality evaluation for whole slide imaging. J Pathol Inform 2012;3:9.
例
>>> from skimage import data >>> from skimage.morphology import disk, ball >>> from skimage.filters.rank import noise_filter >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> out = noise_filter(img, disk(5)) >>> out_vol = noise_filter(volume, ball(5))
- skimage.filters.rank.otsu(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
各ピクセルに対する局所的な大津の閾値です。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
参考文献
例
>>> from skimage import data >>> from skimage.filters.rank import otsu >>> from skimage.morphology import disk, ball >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> local_otsu = otsu(img, disk(5)) >>> thresh_image = img >= local_otsu >>> local_otsu_vol = otsu(volume, ball(5)) >>> thresh_image_vol = volume >= local_otsu_vol
- skimage.filters.rank.percentile(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, p0=0)[source]#
画像の局所パーセンタイルを返します。
局所的なグレイ値分布の下位p0パーセンタイルの値を返します。
パーセンタイル[p0, p1]の間のグレースケール値のみがフィルタで考慮されます。
- パラメータ:
- image2次元配列 (uint8, uint16)
入力画像。
- footprint2次元配列
1と0の2次元配列で表された近傍領域。
- out2次元配列 (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- p0float in [0, …, 1]
パーセンタイル値を設定します。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
- skimage.filters.rank.pop(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
局所的なピクセル数(個体数)を返します。
ピクセル数は、フットプリントとマスクに含まれるピクセル数として定義されます。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
例
>>> from skimage.morphology import footprint_rectangle # Need to add 3D example >>> import skimage.filters.rank as rank >>> img = 255 * np.array([[0, 0, 0, 0, 0], ... [0, 1, 1, 1, 0], ... [0, 1, 1, 1, 0], ... [0, 1, 1, 1, 0], ... [0, 0, 0, 0, 0]], dtype=np.uint8) >>> rank.pop(img, footprint_rectangle((3, 3))) array([[4, 6, 6, 6, 4], [6, 9, 9, 9, 6], [6, 9, 9, 9, 6], [6, 9, 9, 9, 6], [4, 6, 6, 6, 4]], dtype=uint8)
- skimage.filters.rank.pop_bilateral(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, s0=10, s1=10)[source]#
局所的なピクセル数(個体数)を返します。
ピクセル数は、フットプリントとマスクに含まれるピクセル数として定義されます。さらに、ピクセルのグレースケール値は、中心ピクセルのグレースケール値gの区間[g-s0, g+s1]内にある必要があります。
- パラメータ:
- image2次元配列 (uint8, uint16)
入力画像。
- footprint2次元配列
1と0の2次元配列で表された近傍領域。
- out2次元配列 (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- s0, s1int
値の計算に考慮される中心画素のグレイ値を中心とした[s0, s1]区間を定義します。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
例
>>> import numpy as np >>> from skimage.morphology import footprint_rectangle >>> import skimage.filters.rank as rank >>> img = 255 * np.array([[0, 0, 0, 0, 0], ... [0, 1, 1, 1, 0], ... [0, 1, 1, 1, 0], ... [0, 1, 1, 1, 0], ... [0, 0, 0, 0, 0]], dtype=np.uint16) >>> rank.pop_bilateral(img, footprint_rectangle((3, 3)), s0=10, s1=10) array([[3, 4, 3, 4, 3], [4, 4, 6, 4, 4], [3, 6, 9, 6, 3], [4, 4, 6, 4, 4], [3, 4, 3, 4, 3]], dtype=uint16)
- skimage.filters.rank.pop_percentile(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, p0=0, p1=1)[source]#
局所的なピクセル数(個体数)を返します。
ピクセル数は、フットプリントとマスクに含まれるピクセル数として定義されます。
パーセンタイル[p0, p1]の間のグレースケール値のみがフィルタで考慮されます。
- パラメータ:
- image2次元配列 (uint8, uint16)
入力画像。
- footprint2次元配列
1と0の2次元配列で表された近傍領域。
- out2次元配列 (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- p0, p1[0, …, 1] の範囲内の浮動小数点数
値の計算に使用する[p0, p1]パーセンタイル間隔を定義します。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
- skimage.filters.rank.subtract_mean(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
局所平均から引いた画像を返します。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
備考
平均値の減算によりアンダーフローが発生する可能性があります。この潜在的なアンダーフローを補償するために、得られた差分は2の係数でダウンスケールされ、`n_bins / 2 - 1`、つまり局所ヒストグラムの中央値(16ビット画像の場合は`n_bins = max(3, image.max()) + 1`、それ以外の場合は256)だけシフトされます。
例
>>> from skimage import data >>> from skimage.morphology import disk, ball >>> from skimage.filters.rank import subtract_mean >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> out = subtract_mean(img, disk(5)) >>> out_vol = subtract_mean(volume, ball(5))
- skimage.filters.rank.subtract_mean_percentile(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, p0=0, p1=1)[source]#
局所平均から引いた画像を返します。
パーセンタイル[p0, p1]の間のグレースケール値のみがフィルタで考慮されます。
- パラメータ:
- image2次元配列 (uint8, uint16)
入力画像。
- footprint2次元配列
1と0の2次元配列で表された近傍領域。
- out2次元配列 (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- p0, p1[0, …, 1] の範囲内の浮動小数点数
値の計算に使用する[p0, p1]パーセンタイル間隔を定義します。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
- skimage.filters.rank.sum(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
ピクセルの局所合計を返します。
入力配列のデータ型によっては、合計値がオーバーフローする可能性があることに注意してください。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
例
>>> from skimage.morphology import footprint_rectangle # Need to add 3D example >>> import skimage.filters.rank as rank # Cube seems to fail but >>> img = np.array([[0, 0, 0, 0, 0], # Ball can pass ... [0, 1, 1, 1, 0], ... [0, 1, 1, 1, 0], ... [0, 1, 1, 1, 0], ... [0, 0, 0, 0, 0]], dtype=np.uint8) >>> rank.sum(img, footprint_rectangle((3, 3))) array([[1, 2, 3, 2, 1], [2, 4, 6, 4, 2], [3, 6, 9, 6, 3], [2, 4, 6, 4, 2], [1, 2, 3, 2, 1]], dtype=uint8)
- skimage.filters.rank.sum_bilateral(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, s0=10, s1=10)[source]#
フラットカーネル双方向フィルタを適用します。
これは、エッジを保持し、ノイズを低減するノイズ除去フィルターです。画素の空間的な近接性と放射輝度の類似性に基づいて、画素の平均化を行います。
空間的な近接性は、フットプリント(構造要素)によって与えられる局所的な画素近傍のみを考慮することで測定されます。
放射輝度の類似性は、gが現在の画素のグレイレベルである場合、グレイレベル区間[g-s0, g+s1]によって定義されます。
フットプリントに属し、かつこの区間内のグレースケール値を持つピクセルのみが合計されます。
入力配列のデータ型によっては、合計値がオーバーフローする可能性があることに注意してください。
- パラメータ:
- image2次元配列 (uint8, uint16)
入力画像。
- footprint2次元配列
1と0の2次元配列で表された近傍領域。
- out2次元配列 (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- s0, s1int
値の計算に考慮される中心画素のグレイ値を中心とした[s0, s1]区間を定義します。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
例
>>> import numpy as np >>> from skimage import data >>> from skimage.morphology import disk >>> from skimage.filters.rank import sum_bilateral >>> img = data.camera().astype(np.uint16) >>> bilat_img = sum_bilateral(img, disk(10), s0=10, s1=10)
- skimage.filters.rank.sum_percentile(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, p0=0, p1=1)[source]#
ピクセルの局所合計を返します。
パーセンタイル[p0, p1]の間のグレースケール値のみがフィルタで考慮されます。
入力配列のデータ型によっては、合計値がオーバーフローする可能性があることに注意してください。
- パラメータ:
- image2次元配列 (uint8, uint16)
入力画像。
- footprint2次元配列
1と0の2次元配列で表された近傍領域。
- out2次元配列 (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- p0, p1[0, …, 1] の範囲内の浮動小数点数
値の計算に使用する[p0, p1]パーセンタイル間隔を定義します。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
- skimage.filters.rank.threshold(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, shift_z=0)[source]#
画像の局所閾値。
得られた2値マスクは、中心ピクセルのグレースケール値が局所平均値よりも大きい場合にTrueになります。
- パラメータ:
- image([P,] M, N) ndarray (uint8, uint16)
入力画像。
- footprintndarray
1と0のndarrayで表された近傍領域。
- out([P,] M, N) array (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_y, shift_zint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- 戻り値:
- out([P,] M, N) ndarray (入力画像と同じデータ型)
出力画像。
例
>>> from skimage.morphology import footprint_rectangle # Need to add 3D example >>> from skimage.filters.rank import threshold >>> img = 255 * np.array([[0, 0, 0, 0, 0], ... [0, 1, 1, 1, 0], ... [0, 1, 1, 1, 0], ... [0, 1, 1, 1, 0], ... [0, 0, 0, 0, 0]], dtype=np.uint8) >>> threshold(img, footprint_rectangle((3, 3))) array([[0, 0, 0, 0, 0], [0, 1, 1, 1, 0], [0, 1, 0, 1, 0], [0, 1, 1, 1, 0], [0, 0, 0, 0, 0]], dtype=uint8)
- skimage.filters.rank.threshold_percentile(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, p0=0)[source]#
画像の局所閾値。
中心ピクセルのグレイ値が局所パーセンタイル値よりも大きい場合、結果のバイナリマスクはTrueになります。
パーセンタイル[p0, p1]の間のグレースケール値のみがフィルタで考慮されます。
- パラメータ:
- image2次元配列 (uint8, uint16)
入力画像。
- footprint2次元配列
1と0の2次元配列で表された近傍領域。
- out2次元配列 (入力と同じデータ型)
Noneの場合、新しい配列が割り当てられます。
- maskndarray
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- p0float in [0, …, 1]
パーセンタイル値を設定します。
- 戻り値:
- out2次元配列 (入力画像と同じデータ型)
出力画像。
- skimage.filters.rank.windowed_histogram(image, footprint, out=None, mask=None, shift_x=0, shift_y=0, n_bins=None)[source]#
正規化されたスライディングウィンドウヒストグラム
- パラメータ:
- image2次元配列(整数または浮動小数点数)
入力画像。
- footprint2次元配列(整数または浮動小数点数)
1と0の2次元配列で表された近傍領域。
- out2次元配列(整数または浮動小数点数)、オプション
Noneの場合、新しい配列が割り当てられます。
- maskndarray (整数または浮動小数点数), オプション
局所近傍領域に含まれる画像の領域を定義するマスク配列(>0)。Noneの場合、画像全体が使用されます(デフォルト)。
- shift_x, shift_yint、オプション
フットプリントの中心点に追加されるオフセット。シフトはフットプリントのサイズに制限されます(中心は指定されたフットプリント内になければなりません)。
- n_bins整数またはNone
ヒストグラムのビンの数。Noneが渡された場合、
image.max() + 1をデフォルトとします。
- 戻り値:
- out3次元配列(浮動小数点数)
次元が(H,W,N)の配列。ここで(H,W)は入力画像の次元、Nはn_bins、またはパラメータとして値が提供されない場合は
image.max() + 1です。事実上、各ピクセルはN次元特徴ベクトルであり、それはヒストグラムです。特徴ベクトルの要素の合計は1になります(フットプリントとマスクの両方でカバーされたピクセルがウィンドウ内にない場合は、すべての要素が0になります)。
例
>>> from skimage import data >>> from skimage.filters.rank import windowed_histogram >>> from skimage.morphology import disk, ball >>> import numpy as np >>> img = data.camera() >>> rng = np.random.default_rng() >>> volume = rng.integers(0, 255, size=(10,10,10), dtype=np.uint8) >>> hist_img = windowed_histogram(img, disk(5))